用于DNA分析的微流體
1.關于用于DNA分析的微流體的介紹

微流體為生物分析提供了許多優勢:
1.整個生物過程集成并因此簡化了最終用戶
2.高通量,多重和高度平行的分析
3.由于反應時間較短和/或分離時間較短,分析速度更快
4.用于即時應用的便攜式設備
5.試劑消耗低
6.每次分析降低全球成本
所有這些優點已被廣泛用于細胞和分子生物學。本綜述介紹了微流體在后一領域的主要應用及其眾多的DNA分析技術。
2.微流體DNA提取和純化
細胞裂解及其在微流體裝置中的應用
細胞裂解可以通過幾種方法完成:化學裂解,熱裂解,超聲裂解,電裂解,機械裂解。
化學裂解是最常見的實驗室工作臺方法,因為它易于使用而無需特定設備。
該方法可用于片上裂解,但試劑在芯片外儲存,然后通過外部流動系統注入裝置中以進行片上裂解,這使制造復雜化全集成的裂解系統。
熱裂解是PCR實驗室芯片裝置的便利方法,因為裂解所需的熱系統也可用于以下PCR步驟。實際上,先前已經開發了幾種在芯片上集成這些熱裂解和PCR步驟的系統。熱裂解只是將細胞暴露在高溫下。大多數時候,熱電材料集成在芯片上。

來自Privorotskaya等,實驗室芯片,2010
超聲裂解在微流體裝置中呈現的優點,以避免在脈沖模式的局部加熱,從而保持蛋白質結構,同時使一個簡單的積分(不像化學裂解)。
超聲裂解使用超聲波振蕩細胞內的空洞,直到它們崩解。微流體裝置主要使用集成的壓電傳感器來產生這種超聲波。

電裂解使用高電場,其可破壞細胞膜并因此誘導其裂解。由于電極易于集成,電裂解是微流體裝置中常用的技術。然而,該方法存在一些缺點,例如在高壓下可能的水電解并因此形成氣泡。有趣的是,電極也可用于細胞介電電泳以進行上游細胞分選。
來自Medimoon網站
機械裂解可以在微流體裝置中以多種形式進行。可以在微通道內整合尖銳的微納米物體以破壞細胞膜和裂解細胞。通過使用例如高壓地震膜壓縮細胞直至其裂解,也可以使用壓力。也可以使用磁珠通過用旋轉的外部磁體拍打來裂解細胞。在這里可以觀察到,機械裂解需要非常特定的設置,這可能使它們在諸如芯片實驗室系統之類的更完整的設備中的集成變得復雜。
從Quora網站
DNA純化及其在微流體裝置中的應用
大多數微流體DNA純化技術已經適應了在提取柱中制備的宏觀方法,并且在某些緩沖條件下(高度離子和具有適應的pH)使用DNA吸附在二氧化硅珠上。在該DNA吸附后,用洗脫緩沖液(例如水的低離子緩沖液)從二氧化硅顆粒中釋放DNA。
因此,在微流體裝置內,二氧化硅珠可以被機械障礙物或二氧化硅涂覆的磁珠阻擋,以便用磁鐵容易地阻塞這些顆粒用于洗滌和洗脫階段。
其他類型的官能化可用于磁珠(例如核苷酸)。

在毛細血管中使用壓力驅動流動的等速電泳也可用于有效分離DNA。簡而言之,等速電泳使用DNA之間的不同離子遷移率,“慢”和“快”電解質。必須選擇電解質以使DNA具有中間DNA遷移率,并因此在施加電場時聚焦在界面處。甲壓力驅動逆流可用于控制聚焦界面的位置。因此,可以容易地從粗制樣品中提取DNA。
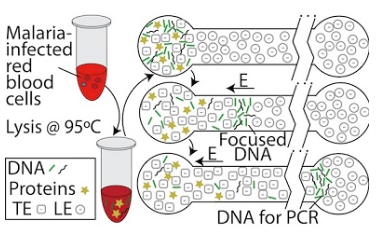
來自Standford微流體實驗室網站
3.微流體DNA電泳
電泳是由均勻電場引起的帶電粒子的位移。基本上,由于DNA由于其糖 - 磷酸骨架而帶負電,因此它將在均勻電場的存在下向陽極電極遷移。具有較高分子量的DNA將較慢遷移,因此可以區分具有不同分子量的DNA。
電泳應用
與較低程度的PCR一樣,DNA電泳分離具有許多應用。
其中一個主要應用是驗證前一段中提到的PCR結果。DNA必須用嵌入劑如溴化乙錠或Sybr Green染色,然后在紫外光下觀察。通過電泳分離,通過大小分離DNA并與校準的分子量梯進行比較以檢查獲得的分子量并將其與預期的PCR結果進行比較。即使分子量得到驗證,分子序列也不會,這使得這種類型的檢測不如熒光探針的qPCR特異性。
電泳也可用于分析通過限制酶切割的DNA分子。這些限制酶在特定的限制性位點切割DNA。然后可以通過電泳分離不同的DNA序列,并根據所用的設備從電泳底物中回收,以將它們用作質粒構建的DNA序列模板(例如,質粒是環狀DNA,通常合成為克隆載體或用于蛋白質)生產)。
電泳也可用于DNA測序。該原則將在下一段中討論。
用于電泳的微流體裝置
電泳最初在凝膠中進行,主要在瓊脂糖(對于較長的DNA)和聚丙烯酰胺(對于較短的DNA)中進行。這些凝膠是多孔的,這解釋了它們根據DNA大小/分子量降低了DNA電泳遷移率。
然后將電泳適應于毛細管,其具有優于凝膠的若干優點。首先,它們能夠使用更高的電壓,從而減少分析時間。其次,由于它們具有大的表面積與體積比,它們有效地消散焦耳熱,這對于不產生溫度梯度以及因此可能干擾電泳效應的對流效應是重要的。電泳遷移率也取決于溫度,因此溫度梯度可能導致遷移區的遷移率和變形的空間依賴性。
隨著微流體技術的出現,DNA電泳成為第一個集成在芯片上的分子過程。這種小型化使得能夠進一步增加處理時間以實現電泳(例如,在幾分鐘內對數百個堿基進行測序),減少試劑消耗并在芯片上組裝其他DNA處理步驟,如前所述。現在,用于DNA電泳的微裝置分析已商業化。值得注意的是,壓力控制也可以與電動力結合使用,以將DNA樣品注入分離通道內。該技術能夠避免具有不同電子遷移率的物種之間的注入差異。
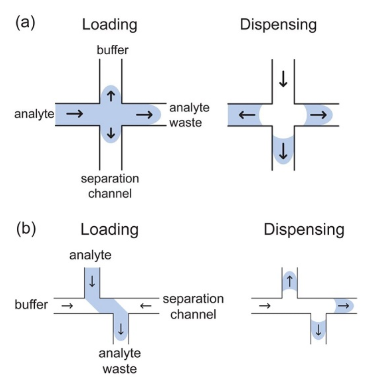
電泳微芯片中的樣品注入方法的實例。(a)浮動注射,(b)雙T模式。改編自Dorfman等,Chemical Review,2012。關于這些注射方法的解釋可以在這篇優秀的評論中找到。
4. DNA測序和微流體
測序是確定DNA序列的核苷酸順序的分析。首次測序是基于Sanger方法開發的。簡而言之, 桑格方法基于PCR方法,但是,代替僅使用常規脫氧核苷酸(dNTP,參見PCR段落),標記的雙脫氧核苷酸(ddNTP)也以低得多的濃度添加。與dNTP不同,ddNTP缺失3'羥基,這意味著當它們在細長DNA鏈的末端添加時,不能繼續伸長。因此,通過與所有不同類型的dNTP(A,T,C,G)和較少的ddNTP進行PCR反應,用不同熒光團標記的ddNTP,具有不同長度的DNA片段和對應于不同核苷酸末端的不同熒光團將是獲得。然后通過電泳鑒別這些DNA片段,并且不同的熒光團將能夠光學區分不同的堿基并因此對DNA進行測序。
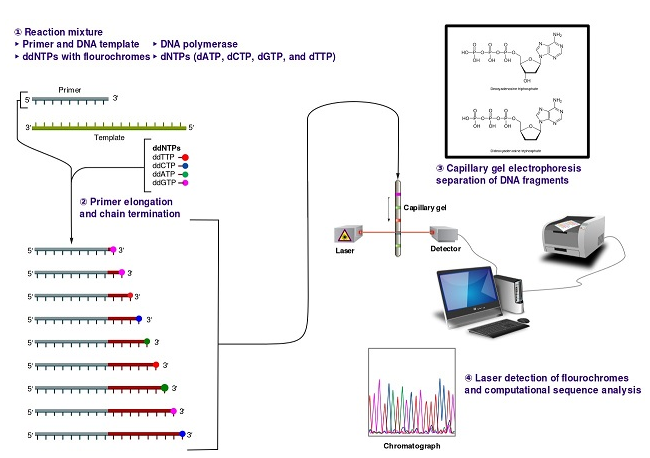
桑格方法原理
微流體已被廣泛用于改善Sanger測序,特別是因為改進電泳和其他測序步驟(DNA純化和PCR)的整合(參見前面的段落)。下一代測序技術對應于能夠實現高通量測序的技術,從而大大降低其成本和持續時間。所開發的系統使用全球微納米技術,生物物理和生物化學技術而不是特別是微流體技術,即使毫無需毫安和微流體來容易地在消耗品內運輸和交換試劑。
在最著名的新一代測序系統中,我們可以通過合成技術,生命技術和使用半導體技術的Ion Torrent命名Illumina及其測序,Pacific Bio使用零模式波導進行單分子實時測序,Roche及其454測序系統使用焦磷酸測序技術。
然而,微流體更直接地用于依賴最初基于微陣列的測序技術的其他系統中,即雜交測序。基本上,待鑒定的序列暴露于大量短標記探針(通常5至10個核苷酸),因此在洗去非雜交探針后光學檢測它們的雜交。研究的序列或探針可以固定在微陣列上。
通過將不同的標記探針置于液滴中并將它們暴露于DNA序列,已經應用了相同的原理。然后通過掃描每個液滴?光學檢測雜交。已經開發了微流體裝置以自動制備測序文庫,即隨機片段化的DNA序列,然后將其用于以下測序步驟。用于自動化文庫制備的裝置現在也由測序公司商業化。
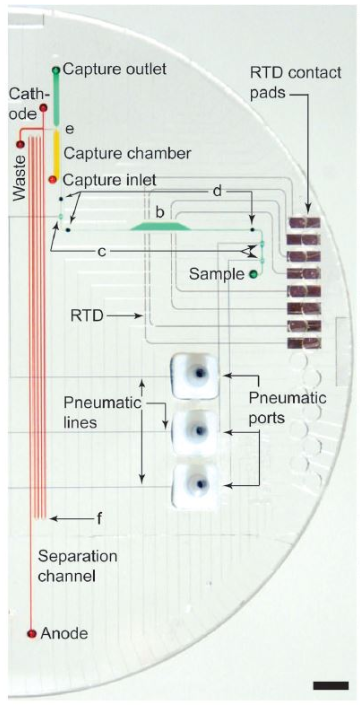
用于Sanger測序的微流體芯片實驗室采集熱循環,樣品純化和芯片電泳。來自Blazej等,PNAS,2006
5.關于用于DNA分析的微流體的結論
我們看到微流體技術在DNA分析中發現了許多重要且關鍵的應用。
由于DNA分析所需的復雜程序和較長的分析時間,微流體技術能夠集成芯片上的所有不同步驟,并大大縮短分析時間。然而,這種技術仍然需要在分子生物學實驗室中被廣泛接受,因為與常規方法相比,其成本目前相對昂貴。因此,它現在更多地用于需要高通量分析或非常靈敏的分析的實驗室。